| 
|
 |

|
| |
"It
has not escaped our notice that the specific pairing we have
postulated immediately suggests a possible copying mechanism
for the genetic material." |
|
6-
La réplication Il
n'a pas échappé à Crick et Watson que le
modèle d'ADN qu'ils avaient établi permettait de
comprendre comment cette molécule peut se reproduire, comment
à partir d'une molécule, on peut facilement en obtenir
deux identiques. On peut lire, d'ailleurs, à la fin de
leur célèbre article dans Nature :
"It
has not escaped our notice that the specific pairing we have
postulated immediately suggests a possible copying mechanism
for the genetic material."
Voyez-vous
à quel mécanisme de copie ils pensaient ? Jetez
un coup d'oeil à l'image ci-dessous. |
|
|
|
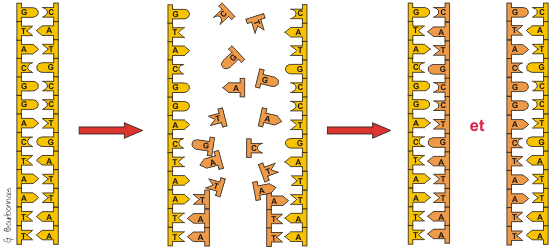
 On
peut facilement séparer les deux brins formant la
molécule d'ADN (les liaisons hydrogènes qui
les relient sont faciles à défaire). À
partir de chacun des deux brins obtenus, on peut reconstituer
la molécule entière en y appariant des nucléotides.
Les nucléotides ne peuvent s'apparier que s'ils sont
complémentaires, A
avec T et C
avec G. Ce faisant, chacun des deux brins
séparés sert en quelque sorte de moule pour
reconstituer l'autre brin. Le noyau de la cellule contient
en tout temps une grande quantité de nucléotides
(des A, T, C et G) pouvant servir à reproduire l'ADN.
Comme nous le verrons plus loin, plusieurs enzymes sont
aussi nécessaires. On
peut facilement séparer les deux brins formant la
molécule d'ADN (les liaisons hydrogènes qui
les relient sont faciles à défaire). À
partir de chacun des deux brins obtenus, on peut reconstituer
la molécule entière en y appariant des nucléotides.
Les nucléotides ne peuvent s'apparier que s'ils sont
complémentaires, A
avec T et C
avec G. Ce faisant, chacun des deux brins
séparés sert en quelque sorte de moule pour
reconstituer l'autre brin. Le noyau de la cellule contient
en tout temps une grande quantité de nucléotides
(des A, T, C et G) pouvant servir à reproduire l'ADN.
Comme nous le verrons plus loin, plusieurs enzymes sont
aussi nécessaires.
À
partir d'une molécule d'ADN et de nucléotides
séparés, on obtient deux molécules
d'ADN parfaitements identiques. Chacune des deux nouvelles
molécules obtenues est formée d'un brin de
la molécule d'origine et d'un nouveau brin assemblé
à partir des nucléotides ajoutés. C'est
ce qu'on a appelé un mode de reproduction semi-conservatif.
|
Voyez-vous
aussi en quoi la structure de l'ADN est particulièrement
bien adaptée à cette fonction de reproduction?
Voir
: La
structure de l'ADN (cliquez sur "Replicate")
|
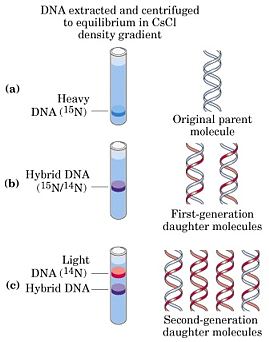 Il
faudra attendre encore cinq ans avant de pouvoir démontrer
expérimentalement ce mode de reproduction. Ce sont
les expériences de Matthew
Meselson et Franklin Stahl qui confirmeront
le modèle semi-conservatif imaginé par Crick
et Watson. Meselson et Stahl faisaient se reproduire des
bactéries dans un milieu contenant des ions ammonium
(NH4+) constitués d'azote 14
(isotope léger) ou d'azote 15 (isotope plus lourd).
Les bactéries fabriquent leurs nucléotides
à partir de ces ions ammonium et les utilisent pour
synthétiser leur ADN. Il
faudra attendre encore cinq ans avant de pouvoir démontrer
expérimentalement ce mode de reproduction. Ce sont
les expériences de Matthew
Meselson et Franklin Stahl qui confirmeront
le modèle semi-conservatif imaginé par Crick
et Watson. Meselson et Stahl faisaient se reproduire des
bactéries dans un milieu contenant des ions ammonium
(NH4+) constitués d'azote 14
(isotope léger) ou d'azote 15 (isotope plus lourd).
Les bactéries fabriquent leurs nucléotides
à partir de ces ions ammonium et les utilisent pour
synthétiser leur ADN.
Expérience
de Meselson et Stahl
Voir aussi: Meselson
and Stahl (cliquez sur Animation et ensuite
sur Narrated)
- Quelles
étaient les trois hypothèses possibles en
ce qui concerne la réplication de l'ADN ?
- Comment
Meselson et Stahl ont-ils mis en évidence le modèle
semi-conservatif ?
Voir
aussi cette animation
Flash (tirée du site Bioanim).
|
|
L'ADN
hélicase
et l'ADN polymérase III
 Dans la cellule, c'est une enzyme du noyau, l'ADN
hélicase, qui sépare l'ADN en
deux brins. L'ADN hélicase agit un peu comme la tirette d'une
fermeture éclair qui sépare les deux parties de la
fermeture.
Dans la cellule, c'est une enzyme du noyau, l'ADN
hélicase, qui sépare l'ADN en
deux brins. L'ADN hélicase agit un peu comme la tirette d'une
fermeture éclair qui sépare les deux parties de la
fermeture.
Les
nucléotides qui serviront à reconstituer le brin
complémentaire sont présents en grande quantité
dans le noyau. La cellule s'en garde toujours une bonne provision.
Chacun de ces nucléotides libres contient trois groupements
phosphates. Lorsqu'ils sont incorporés dans l'ADN, ils
perdent deux phosphates ce qui fournit l'énergie nécessaire
à leur liaison.
C'est
une autre enzyme du noyau, l'ADN
polymérase III qui vient
les apparier un à un sur chacun des deux brins séparés.
L'ADN polymérase III ne peut relier les nouveaux nucléotides
que dans le sens 5' - 3'. La croissance du nouveau brin
se fait donc dans la direction 5' - 3'. Le dessin ci-dessous
serait plus fidèle à la réalité que
le précédent :
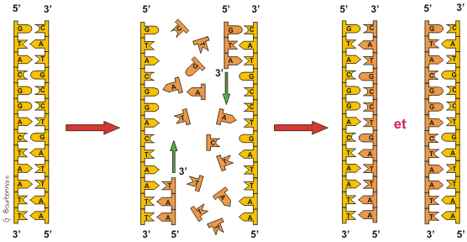
La
croissance du nouveau brin d'ADN se fait dans le sens 5' - 3'
|
Chez
les procaryotes,
une seule ADN hélicase sépare
l'ADN en un point et progresse ensuite tout le long de l'anneau
d'ADN jusqu'à revenir à son point de départ.
Chez
les eucaryotes,
plusieurs ADN hélicases travaillent
simultanément sur un même chromosome. Sur les
plus longs chromosomes, on peut avoir plusieurs centaines
d'hélicases qui travaillent en même temps chacune
à partir d'un point différent d'insertion. |
|
|
Dans
la cellule, l'ADN, après avoir été séparé
en deux brins par l'hélicase, se déroule
sur une certaine longueur. L'ADN polymérase III
assemble alors, au fur et à mesure que la molécule
se sépare, le brin complémentaire 5' - 3'
(celui qui s'apparie au brin d'origine 3' - 5' ).
Lorsqu'une certaine longueur d'ADN a été séparé
en deux brins, une autre ADN polymérase commence à
assembler, dans le sens contraire, le brin complémentaire
de l'autre partie. Pas très clair hein? Regardez plutôt
l'illustration ci-dessous:
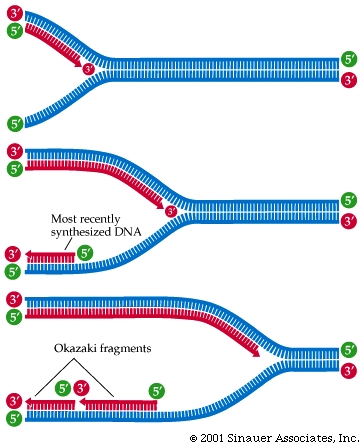
Les
brins nouveaux (en rouge) sont assemblés dans la
direction 5' - 3'.
Sur le brin d'origine 3' - 5' (celui du haut sur cette
image), le nouveau brin s'assemble au fur et à mesure que
l'ADN est séparé en deux brins.
Sur l'autre brin d'origine, le 5 ' -3' (celui du bas
sur l'image), le nouveau brin s'assemble dans la direction contraire
de l'autre nouveau (de droite à gauche sur l'image).
Au fur et à mesure que s'ouvre l'ADN, une ADN polymérase
assemble dans la direction 5' - 3' un court fragment appelé
fragments d'Okazaki.
Le brin d'origine 5' - 3' est donc copié petit
bout par petit bout (chaque petit bout est un fragment d'Okazaki)
par plusieurs ADN polymérases différentes. Les fragments
d'Okazaki ont entre 100 et 200 nucléotides de long chez
les eucaryotes et entre 1000 et 2000 chez les procaryotes.
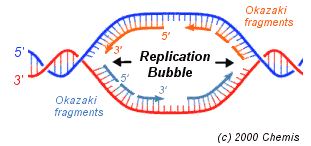
Lorsqu'un chromosome est reproduit, il peut se former des centaines
de sites de réplication comme celui illustré ci-dessus.
|
Petit
calcul |
|
Chez
la bactérie E. coli, la synthèse
du nouveau brin d'ADN se fait à une vitesse d'environ
1500 nucléotides à la seconde.
Sachant que le chromosome de E. coli contient 4,7
millions de paires de bases, combien faut-il de temps, au
minimum, pour reproduire ce chromosome ?
 N.B.
Chez les procaryotes, il n'y a qu'un seul chromosome
et celui-ci est circulaire (l'extrémité
5' est reliée à l'extrémité
3'). La réplication débute en un seul point
du chromosome et elle se fait dans les deux directions
à la fois (vers la droite à partir
de ce point et aussi, en même temps, vers la gauche). N.B.
Chez les procaryotes, il n'y a qu'un seul chromosome
et celui-ci est circulaire (l'extrémité
5' est reliée à l'extrémité
3'). La réplication débute en un seul point
du chromosome et elle se fait dans les deux directions
à la fois (vers la droite à partir
de ce point et aussi, en même temps, vers la gauche).
Votre
résultat doit être compatible avec le fait
qu'une bactérie E.coli peut se reproduire en moins
de 30 minutes lorsque les conditions lui sont favorables.
Donc, vous avez fait une erreur si vous arrivez à
plus de 30 minutes.
|
|
| L'ADN
polymérase III est un complexe enzymatique formé
de plusieurs protéines. Explorez la protéine
bêta de ce complexe en vous rendant à :
E.
coli DNA Polymerase III
Cette unité ressemble à un beigne entourant
le brin d'ADN. Voyez comment s'insère la molécule
d'ADN dans "le trou du beigne" (partie III, Interaction
with DNA ). L'anneau se déplace le long du brin en
assemblant les nucléotides complémentaires.
L'ADN
polymérase des eucaryotes est différente.
Il
existe aussi des polymérases dont la fonction est
de corriger les erreurs qui peuvent se produire lors de
la réplication. Ces enzymes "vérifient"
le travail de l'ADN polymérase III et corrigent toute
erreur que celle-ci pourrait faire (l'ADN polymérase
III peut parfois se tromper en appariant les nouveaux nucléotides;
l'enzyme de correction enlève alors les "mauvais"
nucléotides pour les remplacer par les bons).
Voyez
aussi ces animations qui illustrent le fonctionnement de
l'enzyme:
DNA
Polymerase Beta MOVIES
|
|
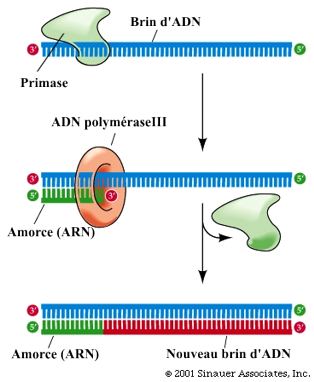
Les amorces
Le dessin
ci-dessus n'est pas encore tout à fait fidèle à
la réalité. Il manque encore quelque chose, les
amorces.
L'ADN polymérase
III ne peut fonctionner que si elle se fixe d'abord sur une amorce.
Cette amorce est formée d'un court segment d'ARN
complémentaire à un segment du brin à copier.
L'amorce mesure environ une dizaine de nucléotides. Partout
où se forme une amorce, une ADN polymérase peut
commencer à assembler des nucléotides.
| L'ARN
(acide ribonucléique), comme nous le verrons plus
loin, est un polymère de ribonucléotides.
Le ribonucléotide est identique au désoxyribonucléotide
à part le fait que son sucre est un ribose plutôt
qu'un désoxyribose (d'où le nom acide
ribonucléique). |
C'est une
autre enzyme, la primase
qui sert à assembler ces amorces. Dès qu'une amorce
est assemblée, l'ADN polymérase III peut commencer
son travail.
À la
fin de la synthèse du brin complémentaire, une enzyme,
l'ADN polymérase I
remplace les ribonucléotides des amorces
par des désoxyribonucléotides (les
A, T, C et G de l'ADN). Le brin d'ARN que formait l'amorce est
donc remplacé par un brin d'ADN.
Une
dernière enzyme, l'ADN ligase
vient rattacher les uns aux autres tous ces segments (les fragments
d'Okazaki et les brins qui ont remplacé les amorces).
C'est
plus facile de visualiser toutes ces étapes en regardant
cette
animation (attention, 10 Mo à télécharger).
|
|
| RECHERCHE
: LA PCR |
|
La
PCR est un processus qui permet de multiplier en de nombreux
exemplaires (on dit amplifier)
un segment d'ADN particulier (la séquence
cible). Cette technique permet d'amplifier
des segments spécifiques à partir d'infimes
échantillons d'ADN (une trace de salive sur un mégot
de cigarette, par exemple, peut suffire).
- Que
veulent dire les lettres PCR ?
- Comment
fonctionne un appareil à PCR ?
- Qu'est-ce
qui permet de sélectionner pour amplicication un
petit segment bien précis d'ADN ?
- Dans
le processus d'amplification, il faut chauffer l'ADN pour
séparer les deux brins (c'est ce qu'on appelle
le point de fusion de l'ADN). À quelle température
les deux brins se séparent-ils?
Vous
pouvez commencer par ces animations :
La
PCR
Présentation
de la PCR
Voir
aussi cette animation
(tirée du site Bioanim).
Si le fichier ne s'exécute pas correctement (c'est
le cas sur mon Firefox; les fichiers Quicktime, c'est vraiment
de la *?&***!!), vous pouvez télécharger
cette version exécutable. |
|

Appareil à PCR
|
©
Gilles Bourbonnais / Cégep de Sainte Foy |
 Dans la cellule, c'est une enzyme du noyau, l'ADN
hélicase, qui sépare l'ADN en
deux brins. L'ADN hélicase agit un peu comme la tirette d'une
fermeture éclair qui sépare les deux parties de la
fermeture.
Dans la cellule, c'est une enzyme du noyau, l'ADN
hélicase, qui sépare l'ADN en
deux brins. L'ADN hélicase agit un peu comme la tirette d'une
fermeture éclair qui sépare les deux parties de la
fermeture.



 On
peut facilement séparer les deux brins formant la
molécule d'ADN (les liaisons hydrogènes qui
les relient sont faciles à défaire). À
partir de chacun des deux brins obtenus, on peut reconstituer
la molécule entière en y appariant des nucléotides.
Les nucléotides ne peuvent s'apparier que s'ils sont
complémentaires, A
avec T et C
avec G. Ce faisant, chacun des deux brins
séparés sert en quelque sorte de moule pour
reconstituer l'autre brin. Le noyau de la cellule contient
en tout temps une grande quantité de nucléotides
(des A, T, C et G) pouvant servir à reproduire l'ADN.
Comme nous le verrons plus loin, plusieurs enzymes sont
aussi nécessaires.
On
peut facilement séparer les deux brins formant la
molécule d'ADN (les liaisons hydrogènes qui
les relient sont faciles à défaire). À
partir de chacun des deux brins obtenus, on peut reconstituer
la molécule entière en y appariant des nucléotides.
Les nucléotides ne peuvent s'apparier que s'ils sont
complémentaires, A
avec T et C
avec G. Ce faisant, chacun des deux brins
séparés sert en quelque sorte de moule pour
reconstituer l'autre brin. Le noyau de la cellule contient
en tout temps une grande quantité de nucléotides
(des A, T, C et G) pouvant servir à reproduire l'ADN.
Comme nous le verrons plus loin, plusieurs enzymes sont
aussi nécessaires. Il
faudra attendre encore cinq ans avant de pouvoir démontrer
expérimentalement ce mode de reproduction. Ce sont
les expériences de Matthew
Meselson et Franklin Stahl qui confirmeront
le modèle semi-conservatif imaginé par Crick
et Watson. Meselson et Stahl faisaient se reproduire des
bactéries dans un milieu contenant des ions ammonium
(NH4+) constitués d'azote 14
(isotope léger) ou d'azote 15 (isotope plus lourd).
Les bactéries fabriquent leurs nucléotides
à partir de ces ions ammonium et les utilisent pour
synthétiser leur ADN.
Il
faudra attendre encore cinq ans avant de pouvoir démontrer
expérimentalement ce mode de reproduction. Ce sont
les expériences de Matthew
Meselson et Franklin Stahl qui confirmeront
le modèle semi-conservatif imaginé par Crick
et Watson. Meselson et Stahl faisaient se reproduire des
bactéries dans un milieu contenant des ions ammonium
(NH4+) constitués d'azote 14
(isotope léger) ou d'azote 15 (isotope plus lourd).
Les bactéries fabriquent leurs nucléotides
à partir de ces ions ammonium et les utilisent pour
synthétiser leur ADN.


 N.B.
Chez les procaryotes, il n'y a qu'un seul chromosome
et celui-ci est circulaire (l'extrémité
5' est reliée à l'extrémité
3'). La réplication débute en un seul point
du chromosome et elle se fait dans les deux directions
à la fois (vers la droite à partir
de ce point et aussi, en même temps, vers la gauche).
N.B.
Chez les procaryotes, il n'y a qu'un seul chromosome
et celui-ci est circulaire (l'extrémité
5' est reliée à l'extrémité
3'). La réplication débute en un seul point
du chromosome et elle se fait dans les deux directions
à la fois (vers la droite à partir
de ce point et aussi, en même temps, vers la gauche).
